shotshop.com
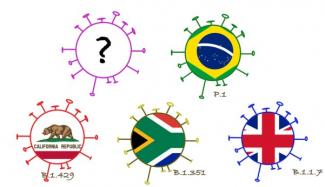
Britische, südafrikanische, brasilianische Varianten: Coronavirus-Mutationen bestimmen aktuell die Nachrichtenlage. Was genau steckt hinter den verschiedenen Varianten?
Dass Viren mutieren können, ist bekannt. Besonders mRNA-Viren wie das Sars-CoV-2 sind anfällig für Mutationen. Seit Beginn der Pandemie gab es weit mehr als 12.000 Mutationen. Viele davon blieben ohne Wirkung. Einige veränderten das Virus aber und führten zu einer Reihe neuer Varianten. Diese Varianten wurden bereits seit Beginn der Pandemie analysiert. In Luxemburg ist das LNS hierfür zuständig. So konnte man zu Beginn anhand der verschiedenen Varianten beobachten, und somit auch ungefähr rückverfolgen, von wo das Virus eingeschleppt wurde (zu Anfang gab es also chinesische Varianten, italienische, spanische etc.) Lange war dies jedoch kein großes Thema, da die verschiedenen Varianten sich nicht wesentlich unterschieden hinsichtlich Ansteckungspotential und Virulenz (also wie schwer sie Menschen krank machen). Forscher haben jedoch seit Anfang der Pandemie davor gewarnt, dass es zu Mutationen kommen könnte, die ansteckender und virulenter seien. Dies ist nun eingetreten. Also zumindest ist gewusst, dass sich zurzeit Mutationen durchsetzen, die ansteckender sind. Ob sie auch virulenter sind, ist noch nicht 100% geklärt, es gibt aber Hinweise darauf. Es hätte auch sein können – wie dies bei Sars-CoV-1 der Fall war (das Virus das verantwortlich war für die Sars-Epidemie 2002) – dass sich eine Virusmutation durchsetzt, die weniger ansteckend oder weniger virulent ist. Es scheint sich allerdings herauszustellen, dass es im Fall von Sars-Cov-2, zumindest zurzeit, in die andere Richtung geht.
Wir gehen im Folgenden mehr im Detail auf die verschiedenen Mutationen ein:
Infobox
Biologisch gesehen sind Viren im Prinzip reines genetisches Erbmaterial – umhüllt von Lipiden und Proteinen (wie z.B. dem Spike-Protein im Falle des Coronavirus). Das genetische Erbmaterial der Viren besteht aus Nukleinsäure-Molekülen, einer Sammlung von Bauanleitungen für verschiedene Proteine. Anders als Menschen, Tiere, Pflanzen oder Bakterien sind Viren aber nicht in der Lage, aus diesen Bauanleitungen selbstständig Proteine, die für die Replikation des Virus nötig sind, herzustellen. Dazu benötigen sie eine Wirtszelle. Haben sie eine solche gekapert, zwingen sie diese dazu, die Virenbauanleitung auszulesen und immer neue Kopien des Virus herzustellen.
Diese Kopiervorgänge laufen allerdings nicht immer sauber ab. Hin und wieder schleicht sich ein Fehler ein, der das Erbmaterial verändert. Eine Mutation entsteht. Manche dieser Kopierfehler bleiben ohne Wirkung. Andere verändern hingegen die Bauanleitung so, dass neue Proteine entstehen. Das bleibt nicht ohne Einfluss auf das Virus. Es kann infektiöser werden oder seinem Wirt größeren Schaden zufügen; es kann dem Immunsystem besser entgehen; es kann sich aber auch besser mit dem Wirt arrangieren. Die Mutationen als solches sind zufällig. Dahinter steckt keine Strategie oder Entwicklungsrichtung. Aus Sicht der Evolution setzen sich allerdings solche Mutationen durch, die eine Vermehrung und Verbreitung des Virus begünstigen. Mit der Zeit entstehen Varianten des Virus, die sich wiederum zu neuen Stämmen entwickeln können.
Desoxyribonukleinsäure, kurz DNA, und Ribonukleinsäure, kurz RNA, sind die Moleküle des Lebens. Beide tragen die Baupläne für alle Proteine, die den Organismus formen – und geben diese an die nächste Generation weiter. Geschrieben werden die Baupläne mit einem Alphabet, das aus lediglich vier „Buchstaben“ besteht: den Basen Adenin, Cytosin, Guanin und Thymin. In der RNA wird anstelle des Thymin übrigens Uracil benutzt. Während die Erbinformation von Mensch und Tier, Pflanze und Pilz, Bakterie und Archaea in der DNA gespeichert ist, liegt sie in einigen Viren – wie zum Beispiel SARS-CoV-2 – in der RNA.
Die Abfolge der Basen in DNA und RNA ist der Code für die Proteine. Der Bauplan des SARS-CoV-2 Virus ist beispielsweise etwa 30.000 Buchstaben lang. Die müssen jedes Mal abgeschrieben werden, wenn das Virus in der Wirtszelle eine Kopie von sich anfertigen lässt. Dabei kommt es zu Schreibfehlern. Mal wird ein Buchstabe vergessen (Deletion). Mal wird er zu viel gesetzt (Insertion). Mal wird ein Buchstabe mit einem anderen vertauscht. Und manchmal auch ein ganzer Abschnitt. Das Ergebnis nennen Wissenschaftler eine Mutation. Und die wird an die nächste Generation weitergegeben.
Mit der Zeit sammeln sich mehr und mehr Mutationen im Virus an. Sind einige davon für die Verbreitung vorteilhaft, dann setzen sich die Viren mit diesem Erbgut gegen ihre nicht mutierten Geschwister durch. Eine neue Variante entsteht. Die ist aber nicht das Ende der Fahnenstange. Denn mit der Zeit kann das Virus so stark mutieren, dass es sich in seiner Funktionsweise deutlich vom Ursprungserreger unterscheidet. Jetzt sprechen Forscher von einem neuen Stamm.
RNA-Viren wie zum Beispiel SARS-CoV-2 mutieren zwar in der Regel häufiger als DNA-Viren wie zum Beispiel das Hepatitis-B-Virus. Doch im Gegensatz zu anderen RNA-Viren wie dem grippeauslösenden Influenza-B-Virus oder dem AIDS auslösenden HI-Virus ist die Mutationsrate bei Coronaviren wiederum wesentlich langsamer. Das kommt daher, dass SARS-CoV-2 und seine Verwandten einen Reparaturmechanismus integriert haben, der Kopierfehler zumindest teilweise korrigiert.
Welche Varianten des Coronavirus SARS-CoV-2 sorgen aktuell für Schlagzeilen?
Aktuell, im Spätwinter 2021, sorgen drei Varianten den SARS-CoV-2 Virus für Schlagzeilen: die britische, die südafrikanische und die brasilianische Variante. Die britische Variante trägt die wissenschaftliche Bezeichnung B.1.1.7, die südafrikanische heißt korrekt B.1.351 und die brasilianische P.1. Amerikanische Forscher gehen außerdem davon aus, dass eine vierte Variante für den starken Anstieg der Neuinfektionen im US-Bundesstaat Kalifornien seit Dezember 2020 verantwortlich ist. Die als CAL.20C bezeichnete Variante wurde zwar schon im Juli 2020 beschrieben, spielte dann aber bis Oktober kaum eine Rolle. Erst Ende des Jahres begann sie, sich stark auszubreiten. (Quelle: Zhang, Wenjuan; Davis, Brian D.; Chen, Stephanie S.; Martinez, Jorge M. Sincuir; Plummer, Jasmine T.; Vail, Eric (2021): Emergence of a novel SARS-CoV-2 strain in Southern California, USA.)
Die Namen „britische Variante“, südafrikanische Variante“ etc. geben nicht gesichert ihre Herkunft an. Sie geben an, wo sie zuerst identifiziert wurden.
Forscher gehen davon aus, dass es auch weiterhin immer wieder zu neuen Mutationen kommen kann, die sich gegen die bisherigen durchsetzen bzw. andere verdrängen.
Was ist über die britische Variante bekannt?
Im Dezember 2020 stiegen die Infektionszahlen in Großbritannien stark an. Ursache war eine neue Variante des SARS-CoV-2 Virus, die den vorläufigen Namen VUI-202012/01 erhielt. Die Abkürzung steht für „variant under investigation“ also „zu prüfende Variante“. Später wurde sie auch als VOC-202012/01 bezeichnet. VOC steht dabei für „variant of concern“ also „Besorgnis erregende Variante“. Besser bekannt ist sie unter der Bezeichnung B.1.1.7.
Zu den wichtigsten Mutationen der britischen Variante gehörten gleich vier Veränderungen an dem Teil des Erbgutes, das die Bauanleitung für das Spike Protein enthält.
Die Zacken der Krone
Spikes sind „die Zacken in der Krone eines Coronavirus“. Diesen winzigen Stacheln auf seiner Oberfläche dienen dem Virus als Andockstelle, um die Wirtszelle zu entern. Die Spikes sind aber auch die Angriffsstelle des Immunsystems, an der das Virus bekämpft wird. Dazu bildet es Antikörper, die die Spikes besetzen. Jetzt kommt das Virus nicht mehr in die Zelle. Es ist neutralisiert.
Zwei Mutationen der britischen Variante – N501Y und K417N genannt – verstärken die Bindung zwischen Virus und Wirtszelle. Eine weitere Mutation E484K genannt – hat das Spike-Protein so verändert, dass manche Antikörper es nun nicht mehr erkennen und besetzen können.
Diese Änderungen sollen dafür gesorgt haben, dass sich die britische Variante leichter verbreitet. Erste mathematische Modelle gingen davon aus, dass sie zwischen 50 und 74 Prozent ansteckender sei. Neuere Abschätzungen der britischen Gesundheitsbehörde geht von einer 25 bis 40-prozentig höheren Infektiosität aus. Die Luxemburger Task-Force-Gruppe für die statistischen Projektionen der Covid-19 Pandemie kommt aktuell auf einen Wert von 39 Prozent. In ihren Modellen hilft ihnen maschinelles Lernen – ein Werkzeug aus dem Bereich der künstlichen Intelligenz – dabei, Muster in den Daten der Gesundheitsbehörde zu finden. Laut Joël Mossong, Epidemiologe an der luxemburgischen Inspection Sanitaire, zeigen die Daten aus dem Contact Tracing in Luxemburg an, dass britische Variante in Luxemburg bis zu 70% ansteckender ist als die herkömmlichen Varianten, dadurch dass die Wahrscheinlichkeit, dass Kontakte positiv werden, gestiegen ist. Fazit ist also, dass hier durchaus noch Unsicherheit besteht. Joël Mossong meint deshalb, um all diese Unsicherheiten zu benennen, dass die britische Variante wohl zwischen 30-80% ansteckender ist.
Nach vorläufigen Erkenntnissen sollten die bisher entwickelten und zugelassenen Impfstoffe auch gegen die britische Variante wirksam sein.
In Luxemburg hat diese Variante mittlerweile die Überhand genommen. In einer repräsentativen Stichprobe gehörten 57,6% der britischen Variante an. Für diese repräsentative Stichprobe wurden 132 Proben genetisch untersucht, was 12% aller positiven Proben dieser Woche entsprach. Von der ECDC, dem Europäischen Zentrum für die Prävention und die Kontrolle von Krankheiten sind 10% vorgesehen, um eine repräsentative Stichprobe zu erhalten.
Was ist über die südafrikanische Variante bekannt?
Auch im Dezember 2020 tauchte in Südafrika eine neue Variante des Virus auf. Sie wird als B.1.351 oder 20H/501Y.V2 bezeichnet. Auch bei dieser sorgen Mutationen – N501Y wie in der britischen Variante und K417N – dafür, dass das Virus fester an die Wirtszelle bindet. Und auch die E484K genannte Mutation, die das Virus vor einigen Antikörpern schützt, ist hier vorhanden. Problematisch bei dieser Variante sind die Hinweise darauf, dass bisher getestete Impfstoffe weniger wirksam sein sollen.Das betrifft in besonderem Maße den Impfstoff des britisch-schwedischen Pharmaunternehmens AstraZeneca. Dem AZD1222 genannten Impfstoff wird in einer südafrikanischen Studie nur eine Wirksamkeit von 21,9 Prozent bescheinigt. Bei der Studie infizierten sich 19 der 750 Teilnehmer trotz Impfstoff mit dem Virus. Unter den 717 Probanden, die ein Placebo erhalten hatten, gab es 23 Infektionen. Allerdings handelte es sich bei allen Erkrankungen um leichte bis mittelschwere Fälle. Die Studie konnte keine Aussage darüber treffen, ob der AstraZeneca-Impfstoff das Risiko eines schweren Krankheitsverlaufes beeinflusst. Ob die südafrikanische Variante ebenfalls leichter übertragbar ist, wird aktuell noch diskutiert.
In der repräsentativen Stichprobe in Luxemburg gehörten 4,5% der positiven Proben der südafrikanischen Variante an.
Was ist über die brasilianische Variante bekannt?
Als 20J/501Y.V3 oder P.1 wird eine Variante bezeichnet, die ebenfalls Ende 2020 in Brasilien gefunden wurde. Auch bei dieser ist der Bauplan für das Spike-Protein verändert, was das Virus ansteckender macht und besser vor dem Immunsystem des Infizierten schützt. Besondere Aufmerksamkeit wurde dieser Variante zuteil, weil sie in Manaus, einer Stadt in Brasilien, zu einer zweiten Welle führte. Nach einem ersten, besonders heftigem Ausbruch gingen Forscher eigentlich davon aus, dass sich bei 76% der Einwohner Antikörper gebildet hätten. Man dachte also, die Herdenimmunität sei eingetreten und eine zweite Welle nicht möglich. Trotzdem brach sie aufgrund der brasilianischen Variante aus.
Infobox
Manaus, die Hauptstadt des brasilianischen Bundesstaates Amazonas, sorgte bereits während der ersten Covid-19 Welle im April 2020 für Schlagzeilen. Damals breitete sich das Virus so schnell aus, dass das Gesundheitssystem der Metropole zusammenbrach. Nach Abklingen der ersten Welle zeigten Tests, dass 76 Prozent der Einwohner Antikörper im Blut trug. Mehr als Dreiviertel der Bevölkerung hatte also Kontakt mit dem Virus und sollte nun zumindest für eine gewisse Zeit immun gegen eine erneute Infektion sein. Außerdem, so die These unter Epidemiologen, sollte damit eine Herdenimmunität erreicht worden sein. Das bedeutet: es sind viele Menschen unempfänglich für einen Erreger, dass es für diesen sehr schwer bis unmöglich wird, sich weiter auszubreiten. Je nach Erreger sollte dieser Punkt erreicht sein, wenn 65 – 70 Prozent der Bevölkerung immunisiert sind. Bei besonders ansteckenden Viren kann aber auch ein deutlich höherer Anteil immuner Menschen in der Bevölkerung nötig sein.
Entgegen der Vermutung, die Herdenimmunität sei erreicht, kam es in Manaus Ende 2020 zu einer zweite Welle, die das Gesundheitssystem der Stadt abermals über seine Grenzen hinaus strapazierte. Die neue Variante, so eine Theorie, könnte dafür verantwortlich sein. Einerseits gehen Forscher davon aus, dass sich die veränderten Viren den Antikörpern des Immunsystems besser entziehen können. Andererseits könnte eine höhere Ansteckungsrate aber auch dafür gesorgt haben, dass das Level für eine Herdenimmunität noch nicht erreicht war Und eine letzte These geht davon aus, dass vielleicht in Wirklichkeit der Anteil an Menschen, die Antikörper hatte, überschätzt wurde. Das könnte an den Hochrechnungen liegen, mit denen die Gesamtanzahl der Menschen mit Antikörpern im Blut aus den Stichproben ermittelt wurden.
Was ist schlimmer: eine ansteckendere Variante oder eine tödlichere?
Wie gefährlich ein Virus ist, hängt von mehreren Faktoren ab. Zwei davon sind die Virulenz – also die Ansteckungsfähigkeit des Virus – und die Letalität – also seine „Tödlichkeit“.
Verursacht eine Variante eine erhöhte Sterblichkeit – wie aktuell bei der britischen Variante diskutiert, aber noch nicht eindeutig nachgewiesen ist – steigt dadurch das Risiko, dem Infekt zu erliegen. Eine höhere Letalität macht das Virus also für den betroffenen Menschen gefährlicher. Eine ansteckendere Variante führt hingegen dazu, dass sich mehr Personen zur gleichen Zeit infizieren. Das führt rein statistisch natürlich auch zu mehr Todesfällen. Außerdem würde es bedeuten, dass mehr Menschen stationär im Krankenhaus behandelt werden müssen, was letztendlich das Gesundheitssystem an seine Grenzen bringen könnte. Sobald aber eine adäquate Behandlung schwerer Fälle durch fehlende Beatmungsgeräte oder mangelnden Sauerstoff nicht mehr möglich ist, steigt ebenfalls die Zahl der Todesopfer. Eine höhere Virulenz könnte demnach laut dem Epidemiologen Adam Kucharski unter Umständen mehr Todesopfer fordern als eine höhere Letalität und macht das Virus deshalb besonders für die Gesellschaft gefährlicher.
Darüber hinaus haben aber noch andere Faktoren einen Einfluss. So gehen einige Wissenschaftler gehen davon aus, dass die britische Variante auch zu einem längeren Krankheitsverlauf führt. Dadurch würde sich der Zeitraum verlängern, in denen ein Covid-19 Patient andere Menschen anstecken kann. Das wiederum würde das Gesundheitssystem vor die gleichen Herausforderungen stellen, wie eine höhere Infektiosität. Anderseits wird aber diskutiert, dass die neuen Varianten allgemein mit einem stärkeren Krankheitsverlauf einhergehen und so die Zahl der asymptomatischen Fälle – also Fälle mit leichterem, unauffälligem Krankheitsverlauf – zurückgeht. Dieser Effekt würde wiederum helfen, die Pandemie besser einzugrenzen. Denn weniger symptomlose oder schwach ausgeprägte Krankheitsverläufe bedeuten weniger versteckte Infektionsketten und damit eine bessere Nachverfolgung.
Welchen Effekt haben diese Varianten auf den Pandemieverlauf?
Allgemein gehen Wissenschaftler davon aus, dass die neuen Varianten den Pandemieverlauf wieder verschärfen könnten. So breitete sich die neue Variante in Großbritannien trotz Lockdown-Maßnahmen stärker aus. Die Reproduktionszahl – also wie viele Menschen eine infizierte Person im Durchschnitt ansteckt – bezifferten Wissenschaftler aktuell auf 1,4. Bei der bisherigen Form des Virus liegt sie zurzeit bei 0,9. Eine ansteckendere Variante führt aber auch dazu, dass die Herdenimmunität erst später erreicht wird, also mehr Menschen durch überstandene Krankheit oder durch Impfung immunisiert sind.
In Luxemburg beschäftigt sich die Task-Force-Gruppe für die statistischen Projektionen mit der Vorhersage des zukünftigen Infektionsgeschehens. Dabei setzen die Wissenschaftler auf ein Modell, das aus aktuellen Infektionsdaten mithilfe von Methoden aus dem Bereich des maschinellen Lernens den weiteren Verlauf der Pandemie abschätzt. Ohne Berücksichtigung der britischen Variante ging das Modell in der zweiten Hälfte Februar von weiter sinkenden Fallzahlen aus. Wird die britische Variante aber berücksichtigt, so zeichnete sich ein Wendepunkt für Ende Februar ab, mit ab dann wieder steigenden Fallzahlen. Würden die sozialen Interaktionen auf dem aktuellen Level weitergeführt, dann erwarten die Forscher den nächsten Peak der Pandemie im Mai. Der Prognose nach würde dieser ähnlich stark ausfallen wir im Oktober 2020. Mit etwa 600 Fällen pro Tag für Luxemburg gelangte das Gesundheitssystem damals fast an seine Schmerzgrenze.
Allerdings sind in diesen Modellen die Szenarien so berechnet, dass das Verhalten der Gesellschaft sich nicht verändert. Wenn die Lage sich aber zuspitzt, verändern die Menschen in der Regel ihr Verhalten – oder die Politik beschließt neue Maßnahmen. So dass mittel- oder langfristige Modellrechnungen mit großen Unsicherheiten behaftet sind. Sie geben an, was passieren würde, wenn es so weitergehen würde, wie die Tendenz der letzten Tage. In anderen Worten: Sie sind nicht als Vorhersagewerkzeug gedacht. Sondern sie geben die Tendenz für die nächsten Tage und Wochen an, wenn die Tendenz der letzten Tage so weiterläuft. Sie sind ein nützlicher Richtwert. Aber keine Wahrsagerei. Hier kommt es oftmals zu Missverständnissen in der öffentlichen Debatte.
Was begünstigt Mutationen und könnte man sie vielleicht sogar aufhalten?
Wie bereits am Anfang des Artikel gesagt, sind Mutationen zufällige Ereignisse, die nicht unbedingt einen Effekt auf das Virus und seine Funktion haben. Sie treten mit einer gewissen Wahrscheinlichkeit auf, sobald das Virus in der Wirtszelle kopiert wird. Das bedeutet: Je mehr Menschen vom Virus befallen sind, umso öfter wird es kopiert und umso öfter können Fehler in seinem Bauplan entstehen. Erweisen sich nun einige dieser Mutationen als günstig für das Virus, können sie sich als Variante durchsetzen und die ursprüngliche Form verdrängen.
Günstig ist hierbei alles, was die Verbreitung des Virus fördert. Dazu zählen eine höhere Infektiosität ebenso wie eine bessere Tarnung vor dem Immunsystem. Wie häufig sich neue Varianten erfolgreich etablieren und in welche Richtung sie dabei gehen, hängt von ihrer Umwelt ab. Im Körper ist das vor allem das Immunsystem. Dessen Aufgabe ist es, das Virus schnell zu eliminieren. Zufällig entstandenen Varianten, die Angriffen des Immunsystems entgehen, können sich deshalb eher verbreiten und die Ursprungsformen ersetzen. Das könnte dazu führen, dass neue Varianten auftreten, die zumindest bestimmten Angriffen des Immunsystems entgehen. Allerdings verfügt das Immunsystem über mehrere Waffen und passt sich seinerseits an die veränderten Viren an.
Mutationen gänzlich zu verhindern, ist nicht möglich. Und auch die Idee, bestimmte Varianten an ihrer Verbreitung zu hindern, hat sich als praktisch nicht umsetzbar erwiesen.
Wie ist die Wahrscheinlichkeit, dass sich in Zukunft noch schlimmere Varianten entwickeln könnten – oder aber das Virus sich abschwächt?
Nach aktuellem Kenntnisstand handelt es sich beim SARS-CoV-2 Virus um eine Zoonose. So nennen Wissenschaftler einen Krankheitserreger, der normalerweise in Tieren vorkommt und nur versehentlich auf den Menschen übergesprungen ist. Beim SARS-CoV-2 Virus gelten Fledermäuse als die wahrscheinlichsten Virenwirte. Tiere, die schon lange mit einem solchen Erreger existieren, haben eine gewisse Koexistenz entwickelt. Sie tragen das Virus zwar in sich, erkranken in der Regel aber nicht.
Verirrt sich der Erreger nun in einen Menschen, sieht das anders aus. Für das Virus sind wir fremdes Terrain und für unser Immunsystem ist das Virus unbekannt. Das kann schnell eskalieren. Neue Zoonosen münden deshalb oft in schweren Krankheitsverläufen und enden häufiger tödlich. Das liegt mitunter auch daran, dass das Immunsystem über das Ziel hinausschießt und – wie bei COVID-19 der Fall – schwere Entzündungsreaktionen im Körper hervorruft. Mit der Zeit aber lernen sich Eindringling und neuer Wirt besser kennen. Das Immunsystem schärft seine Waffen und vom Virus bleiben jene Varianten, die diesen entgehen. Das Virus wird heimisch, schadet seinem Wirt in der Regel aber weniger.
Tatsächlich gab es bereits zu Anfang der Pandemie Anzeichen für Varianten, die mit einem milderen Krankheitsverlauf in Verbindung gebracht wurden. Allerdings wiesen die Wissenschaftler damals darauf hin, dass es sich lediglich um lokale Varianten handelte. Die vorherrschende Virusform hätten sie bisher nicht verdrängt. (Quellen 22 und 23 in Infobox)
Autoren: Kai Dürfeld (scienceRELATIONS – Wissenschaftskommunikation), Jean-Paul Bertemes (FNR)
Infobox
- Roossinck, Marilyn J. (2020): Viren! Helfer, Feinde, Lebenskünstler - in 101 Porträts. 2. Auflage 2020. Berlin: Springer Berlin; Springer.
- Corum, Jonathan; Zimmer, Carl (2021): Coronavirus Variants and Mutations. The New York Times. Online verfügbar unter https://www.nytimes.com/interactive/2021/health/coronavirus-variant-tracker.html?campaign_id=34&emc=edit_sc_20210209&instance_id=26918&nl=science-times®i_id=75221209&segment_id=51277&te=1&user_id=7dfb557d6b820bd9edaad4dca10d0931, zuletzt aktualisiert am 15.02.2021, zuletzt geprüft am 17.02.2021.
- Sanjuán, Rafael; Nebot, Miguel R.; Chirico, Nicola; Mansky, Louis M.; Belshaw, Robert (2010): Viral mutation rates. In: Journal of virology 84 (19), S. 9733–9748. DOI: 10.1128/JVI.00694-10.
- Thal, Dana (2021): Mutationen – ein Tauziehen zwischen Virus und Wirt. Nationale Forschungsplattform für Zoonosen. Online verfügbar unter https://zoonosen.net/mutationen-ein-tauziehen-zwischen-virus-und-wirt, zuletzt aktualisiert am 17.02.2021, zuletzt geprüft am 21.02.2021.
- Zhang, Wenjuan; Davis, Brian D.; Chen, Stephanie S.; Martinez, Jorge M. Sincuir; Plummer, Jasmine T.; Vail, Eric (2021): Emergence of a novel SARS-CoV-2 strain in Southern California, USA.
- Mölling, Karin (2020): Viren. Supermacht des Lebens. 1. Auflage. München: C.H.Beck.
- Volz, Erik; Mishra, Swapnil; Chand, Meera; Barrett, Jeffrey C.; Johnson, Robert; Geidelberg, Lily et al. (2021): Transmission of SARS-CoV-2 Lineage B.1.1.7 in England: Insights from linking epidemiological and genetic data. DOI: 10.1101/2020.12.30.20249034.
- Davies, Nicholas G.; Abbott, Sam; Barnard, Rosanna C.; Jarvis, Christopher I.; Kucharski, Adam J.; Munday, James et al. (2020): Estimated transmissibility and severity of novel SARS-CoV-2 Variant of Concern 202012/01 in England. In: medRxiv, 2020.12.24.20248822. DOI: 10.1101/2020.12.24.20248822.
- Public Health England (2021): Investigation of novel SARS-CoV-2 variant. Variant of Concern 202012/01. Technical briefing 5. Public Health England. Online verfügbar unter https://assets.publishing.service.gov.uk/government/uploads/system/uploads/attachment_data/file/959426/Variant_of_Concern_VOC_202012_01_Technical_Briefing_5.pdf, zuletzt geprüft am 22.02.2021.
- Tertilt, Mathias; Gerhard, Saskia (2021): Corona: Wie gefährlich sind die neuen Mutationen? Neue Virusvarianten. Online verfügbar unter https://www.quarks.de/gesundheit/medizin/corona-wie-gefaehrlich-sind-die-neuen-mutationen/, zuletzt aktualisiert am 26.01.2021, zuletzt geprüft am 08.02.2021.
- Greaney, Allison J.; Loes, Andrea N.; Crawford, Katharine H.D.; Starr, Tyler N.; Malone, Keara D.; Chu, Helen Y.; Bloom, Jesse D. (2021): Comprehensive mapping of mutations to the SARS-CoV-2 receptor-binding domain that affect recognition by polyclonal human serum antibodies. DOI: 10.1101/2020.12.31.425021.
- Wang, Zijun; Schmidt, Fabian; Weisblum, Yiska; Muecksch, Frauke; Barnes, Christopher O.; Finkin, Shlomo et al. (2021): mRNA vaccine-elicited antibodies to SARS-CoV-2 and circulating variants. In: bioRxiv : the preprint server for biology. DOI: 10.1101/2021.01.15.426911.
- Madhi, Shabir A.; Baillie, Vicky; Cutland, Clare L.; Voysey, Merryn; Koen, Anthonet L.; Fairlie, Lee et al. (2021): Safety and efficacy of the ChAdOx1 nCoV-19 (AZD1222) Covid-19 vaccine against the B.1.351 variant in South Africa. DOI: 10.1101/2021.02.10.21251247.
- Tegally, Houriiyah; Wilkinson, Eduan; Giovanetti, Marta; Iranzadeh, Arash; Fonseca, Vagner; Giandhari, Jennifer et al. (2020): Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. DOI: 10.1101/2020.12.21.20248640.
- Buss, Lewis F.; Prete, Carlos A.; Abrahim, Claudia M. M.; Mendrone, Alfredo; Salomon, Tassila; Almeida-Neto, Cesar de et al. (2021): Three-quarters attack rate of SARS-CoV-2 in the Brazilian Amazon during a largely unmitigated epidemic. In: Science (New York, N.Y.) 371 (6526), S. 288–292. DOI: 10.1126/science.abe9728.
- Fine, Paul; Eames, Ken; Heymann, David L. (2011): "Herd immunity": a rough guide. In: Clinical infectious diseases : an official publication of the Infectious Diseases Society of America 52 (7), S. 911–916. DOI: 10.1093/cid/cir007.
- Sabino, Ester C.; Buss, Lewis F.; Carvalho, Maria P. S.; Prete, Carlos A.; Crispim, Myuki A. E.; Fraiji, Nelson A. et al. (2021): Resurgence of COVID-19 in Manaus, Brazil, despite high seroprevalence. In: The Lancet 397 (10273), S. 452–455. DOI: 10.1016/S0140-6736(21)00183-5.
- Faria, Nuno R. (2021): Genomic characterisation of an emergent SARS-CoV-2 lineage in Manaus: preliminary findings. Online verfügbar unter https://virological.org/t/genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-manaus-preliminary-findings/586, zuletzt aktualisiert am 12.01.2021, zuletzt geprüft am 21.02.2021.
- https://twitter.com/adamjkucharski/status/1343567425107881986?lang=de
- Hintergrundgespräch mit Dr. Alexander Skupin, Associate professor/Chief scientist 2 in Modelling of Biomedical Data am LCSB der Universität Luxemburg und Leiter der Task-Force-Gruppe für die statistischen Projektionen in der Covid-19 Pandemie
- Sun, Xinjuan; Wang, Tianyuan; Cai, Dayong; Hu, Zhiwei; Chen, Jin’an; Liao, Hui et al. (2020): Cytokine storm intervention in the early stages of COVID-19 pneumonia. In: Cytokine & Growth Factor Reviews 53, S. 38–42. DOI: 10.1016/j.cytogfr.2020.04.002.
- Toyoshima, Yujiro; Nemoto, Kensaku; Matsumoto, Saki; Nakamura, Yusuke; Kiyotani, Kazuma (2020): SARS-CoV-2 genomic variations associated with mortality rate of COVID-19. In: Journal of human genetics 65 (12), S. 1075–1082. DOI: 10.1038/s10038-020-0808-9.
- Young, Barnaby E.; Fong, Siew-Wai; Chan, Yi-Hao; Mak, Tze-Minn; Ang, Li Wei; Anderson, Danielle E. et al. (2020): Effects of a major deletion in the SARS-CoV-2 genome on the severity of infection and the inflammatory response: an observational cohort study. In: The Lancet 396 (10251), S. 603–611. DOI: 10.1016/S0140-6736(20)31757-8.