shotshop.com
Chercheur dans un laboratoire de niveau de sécurité P3. Elle porte un appareil filtrant l'air.
Le coronavirus maintient actuellement le monde en haleine. Jusqu`à présent (état du 29 janvier 2020), il y a 7783 cas confirmés de personnes infectées dans 20 pays (7678 en Chine) et 170 personnes sont décédées en Chine suite à la maladie respiratoire déclenché par le virus. En même temps, le virus occupe des chercheurs qui travaillent en urgence pour obtenir rapidement plus d’informations sur le nouveau pathogène.
Quelles analyses sont effectués par les chercheurs pour obtenir quel type d’informations? Et pour développer des traitements ou des vaccins?
Explications par Dr Chantal Snoeck, virologue au sein du Luxembourg Institute of Health (LIH).
Dr Snoeck, vous travaillez au LIH sur le virus de la grippe porcine H1N1 parmi d’autres, qui était responsable pour une pandémie globale en 2009. Comment les scientifiques identifient-ils un nouveau virus contagieux comme le coronavirus ?
CS : Différentes étapes sont possibles pour l’identification d’un nouveau virus. Dans le cas du nouveau coronavirus, ce sont d’abord des données épidémiologiques qui ont permis d’éveiller les soupçons des autorités sanitaires que quelque chose d’anormal était en train de se dérouler : plusieurs patients hospitalisés dans la ville de Wuhan souffraient de pneumonie dont la cause n’était pas connue.
L’inefficacité des traitements antibiotiques ainsi que les caractéristiques observées lors des radiographies des poumons pointaient vers une pneumonie d’origine virale et non bactérienne. A partir de cette notification, une enquête épidémiologique chez les patients atteints a permis d’identifier un marché aux poissons comme facteur d’exposition commun entre ces différents patients. »
Et comment est-ce que l’identité précise du nouveau pathogène est alors déterminée ?
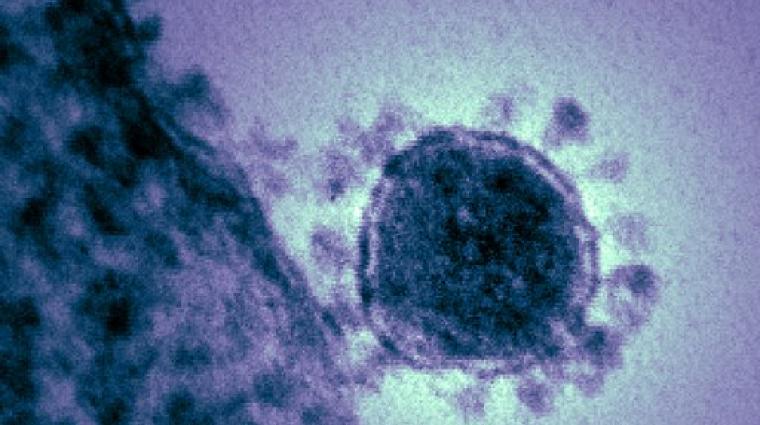
CS : L’identité du pathogène peut être élucidée notamment grâce à la microscopie électronique : les caractéristiques morphologiques d’un virus sont indicatives de la famille à laquelle il appartient. Notamment, les coronavirus tirent leur nom de leur forme sphérique entourée d’une couronne – les glycoprotéines ancrées dans la membrane du virus et essentielles pour l’attachement et l’entrée du virus dans la cellule qu’il infecte.
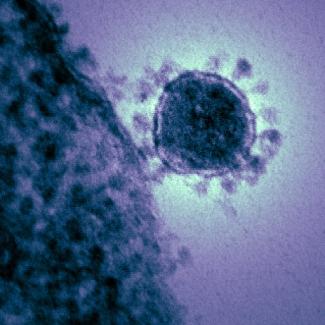
Image: Une micrographie électronique à transmission colorisée du coronavirus lié au syndrome respiratoire du Moyen-Orient (MERS-Cov) qui a apparu en 2012.
Depuis quelques années, une nouvelle technique complémente la panoplie de tests de laboratoire disponibles pour l’identification et la caractérisation d’un agent pathogène et révolutionne l’épidémiologie dite « moléculaire »: le séquençage à haut débit – Next Generation Sequencing en anglais.
A partir d’une culture de virus, voire même de l’échantillon du patient, il est désormais possible d’obtenir la séquence complète du génome viral en quelques heures. Par exemple, le séquençage du génome du SRAS coronavirus a duré 1 mois après l’identification de son agent causal en 2002/2003, alors que le génome du nouveau coronavirus était déjà disponible sur la base de données génétiques GISAID le 10 janvier, quelques jours seulement après l’identification d’un coronavirus.
Infobox
Le séquençage du matériel génétique est possible depuis 1977 grâce à F. Sanger et collègues, mais nécessite la pré-amplification d’une petite portion du génome grâce à des amorces spécifiques, càd des petits brins d’ADN dont on connait la séquence et qui sont complémentaires du fragment d’ADN cible. Cette technique est donc limitante à la fois par la taille des fragments séquencés, le nombre de séquences obtenues ainsi que la connaissance a priori du génome. Et c’est précisément sur ces derniers aspects que le séquençage à haut débit a révolutionné le séquençage : l’information a priori n’est plus nécessaire, et on obtient une quantité massive de séquences qui se chevauchent, permettant ensuite de reconstruire un génome par des outils bioinformatiques.
En effet, c’est impressionnant. Comment le degré de relation avec d'autres virus est-il alors déterminé?
CS : La séquence du génome du nouveau coronavirus est comparée avec le génome des autres coronavirus connus : le degré de relation, aussi appelé la distance génétique, entre deux virus correspond au pourcentage de bases identiques entre ces deux virus tout au long de leur génome, calculé grâce à des modèles statistiques qui prennent en compte l’évolution génétique des organismes.
De plus, les analyses phylogénétiques permettent de retracer les liens de parenté et d’évolution entre des séquences génétiques, ici les génomes de différents coronavirus, grâce aux signaux que les séquences contiennent. Ces résultats sont représentés sous forme d’un arbre qui résume les liens probables, l’histoire génétique. On peut comparer cela à la construction d’un arbre généalogique.
Quelles conclusions les chercheurs ont-ils pu tirer quant au nouveau coronavirus ?
CS : Dans le cas du 2019-nCoV, des analyses phylogénétiques des séquences obtenues jusqu’à présent montrent que tous les cas ont un ancêtre commun récent, càd qu’ils proviennent très certainement d’une nouvelle infection qui s’est ensuite transmise d’homme à homme. Elles permettent aussi d’estimer de quand date cet ancêtre commun : probablement en novembre 2019, et étayent les données épidémiologiques classiques, par exemple pour retracer les contacts entre les cas.
Une semaine après le génome du nouveau coronavirus, un test de diagnostic était également disponible. Comment se développe un tel test de diagnostic?
CS : De nos jours, la présence d’un virus dans un échantillon peut être rapidement détectée par la technique de réaction de polymérisation en chaîne (PCR). Cette technique se base sur l’amplification d’une portion du génome du virus s’il est présent dans l’échantillon grâce à des amorces spécifiques au génome du virus d’intérêt. La publication rapide du génome du 2019-nCoV a été instrumentale pour le développement rapide de PCRs pour sa détection.
Pour développer un tel test, il suffit de trouver une région du génome du virus qui répond à certains critères tels qu’être suffisamment différente des autres coronavirus pour s’assurer que le test ne va pas les détecter aussi. L’identification de cette région se fait par le biais d’outils bioinformatiques comparant les séquences des génomes viraux. Ensuite, le test est validé, entre autre, en testant des échantillons dont on connait le statut infectieux (positif ou négatif pour le virus) et en vérifiant qu’il ne détecte pas d’autres virus apparentés (d’autres coronavirus) ou qui causent des symptômes similaires (par exemple le virus de la grippe).
Quelles autres analyses sont ensuite effectués par les chercheurs pour obtenir rapidement plus d'informations sur le nouveau virus?
CS : Contrairement au virus Ebola qui doit être manipulé dans un laboratoire de niveau de sécurité 4 en nombre limité dans le monde, le nouveau coronavirus peut être manipulé dans un laboratoire de niveau 3, ce qui va faciliter sa caractérisation expérimentale. Après sa découverte, le virus doit être caractérisé du mieux possible in vitro (au laboratoire, en dehors d’un organisme vivant) et in vivo (dans des organismes vivants) afin de le comprendre.
Les cellules ciblées par le virus peuvent être déterminées en essayant d’infecter un panel de lignée cellulaire d’origines animale et humaine. Cela permet aussi d’investiguer le spectre d’hôte potentiel du virus, afin de déterminer son origine potentielle ainsi que les autres animaux susceptibles de maintenir le cycle de transmission du virus.

Cette photographie de 2005 représente l’un des microbiologistes du personnel du Centers for Disease Control and Prevention examinant un virus de la grippe, contenu dans un flacon calibré contenant un milieu de culture surnageant. La photo est prise dans un environnement de laboratoire de niveau de biosécurité 3, où les scientifiques travaillent sous une hotte à flux, où l'air à l'extérieur de la hotte est aspiré dans les limites de la hotte et est ensuite filtré de tout agent pathogène avant d'être recirculé à l'intérieur de l'atmosphère de laboratoire autonome. Les recommandations actuelles pour le travail avec le nouveau coronavirus sont en fait de travailler dans un environnement de niveau de biosécurité 2, sauf pour les cas exceptionnels de l’isolement du virus en culture cellulaire et la caractérisation initiale des agents viraux récupérés dans les cultures d'échantillons 2019-nCoV, qui doivent être fait au niveau de biosécurité 3. Photo: James Gathany Content Providers(s): CDC
Et au niveau moléculaire ?
CS : Le récepteur cellulaire, càd la molécule avec laquelle le virus interagit pour pouvoir rentrer dans la cellule cible, a également déjà été identifié in vitro. Cette connaissance permet ensuite le développement potentiel de molécules pour bloquer son entrée dans la cellule. Dans ce contexte, la bioinformatique est également devenue un outil essentiel dans la caractérisation des pathogènes. Par exemple, une analyse modélisant de la structure de la glycoprotéine du nouveau coronavirus, se basant sur le virus SRAS, suggère que le récepteur cellulaire du virus pourrait être identique au virus SRAS ainsi que d’autres coronavirus.
La compréhension du mode de réplication du virus dans la cellule est aussi importante pour le développement d’une molécule qui pourrait diminuer sa réplication ou sa sortie de la cellule infectée, comme c’est le cas de l’oseltamivir pour le virus de la grippe. Le développement d’un anticorps monoclonal ciblant spécifiquement le virus est une autre avancée possible, comme le Palivizumab qui peut être administré pour prévenir les cas sévères du virus respiratoire syncytial chez les très jeunes enfants.
Il semble qu’on peut déjà déterminer beaucoup dans le laboratoire ou avec l’ordinateur. Est-ce que des expériences animales sont alors encore nécessaire ?
CS : L’expérimentation animale reste importante, notamment pour comprendre le mode de transmission exact d’un virus, son degré de transmission entre un animal infecté et un animal sain, les facteurs qui peuvent influencer la sévérité de l’infection ou pour tester des traitements expérimentaux avant les essais cliniques chez l’homme.
Dans le cas du virus de la grippe par exemple, le furet est considéré comme modèle expérimental reproduisant le mode de transmission du virus et est utilisé pour évaluer la capacité. Cependant les modèles animaux pour les coronavirus tels que SRAS et MERS sont difficiles à établir et ont des limitations.
En parallèle, des études séro-épidémiologiques chez les animaux (déterminer quel animal a des anticorps contre ce virus particulier) devront complémenter les études in vitro/in vivo pour comprendre l’étendue du spectre d’hôte du virus. De telles études chez l’homme, chez des personnes ayant des contacts fréquents avec ses animaux, permettront aussi de déterminer si des infections antérieures et non détectées pourraient avoir eu lieu.
Les options de traitement ou des vaccins, sont-ils également testés ou développés immédiatement?
CS : Au vu des symptômes comparables entre ceux induits par SRAS et le 2019-nCoV et l’existence d’antiviraux, des scientifiques se sont déjà basés sur ces connaissances pour traiter de manière expérimentale certains patients infectés par le 2019-nCoV avec lopinavir et ritonavir, des inhibiteurs de protéases notamment utilisés dans le traitement du VIH . D’autres traitements expérimentaux développés contre SRAS ou MERS coronavirus pourraient potentiellement aider dans la lutte contre le nouveau coronavirus.
Plus rapidement que jamais, le développement de vaccins a déjà commencé et 3 projets basés sur 3 méthodologies différentes sont déjà supportés financièrement. Encore une fois, la disponibilité de la séquence du génome viral a contribué au lancement rapide des essais expérimentaux, ceci combiné aux avancées technologiques en matière de vaccins recombinants. La recherche pour un vaccin contre le MERS CoV permettra également d’accélérer l’obtention d’un vaccin contre 2019-nCoV.
Quelles sont les prochaines étapes pour les chercheurs travaillant sur le nouveau coronavirus?
- La compréhension de son mode de transmission exacte : par les gouttelettes (donc contamination par contact proche) ou par aérosols (contamination entre deux personnes plus éloignées possible)
- La compréhension du spectre de symptômes induits par le virus, la proportion réelle de cas légers, voire asymptomatiques, par rapport aux cas plus sévères plus facile à identifier
- La recherche du réservoir animal du virus
- La détermination du spectre d’hôtes
- La compréhension du mode de réplication exact du virus
- La recherche d’un traitement efficace
- Le développement d’un vaccin
Dr Snoeck, un Grand Merci pour ces précisions très détaillées !
Chantal Snoeck est virologue au sein du l’équipe de virologie clinique et appliquée, dans le département infection et immunité au LIH. Les activités du groupe comprennent la surveillance, le diagnostic, les enquêtes sur les épidémies, l'épidémiologie moléculaire et l'immunité contre diverses maladies virales telles que la rougeole. Les informations sur la prévalence, la variabilité, la pathogénicité des virus et le degré d’immunité de la population sont importantes pour les décideurs en santé publique pour contrôler et gérer les maladies infectieuses. Chantal Snoeck travaille plus particulièrement sur les maladies virales zoonotiques telles que la grippe aviaire et porcine, le virus de l’hépatite E, ainsi que les maladies animales virales à forte conséquence économique.
Texte: Chantal Snoeck (LIH)
Questions et édition: Michèle Weber (FNR)
Photo: Public Health Image Library du CDC.
Infobox
Gralinski and Menachery. Return of the coronaviruses: 2019-nCoV. Viruses 2020.