shotshop.com
Forscher in einem BL3-Sicherheitslabor. Sie trägt ein Atemfiltergerät.
Das neue Coronavirus aus China hält derzeit die Welt in Atem. Bisher (Stand 29. Januar 2020) gibt es 7.783 bestätigte Fälle von Infektionen in 20 Ländern (7.678 in China), und 170 Menschen sind in China an den Folgen einer durch das Virus ausgelösten Atemwegserkrankung gestorben. Gleichzeitig beschäftigt das Virus die Forschern, die emsig daran arbeiten, schnellstmöglich mehr Informationen über den neuen Erreger zu erhalten.
Welche Analysen werden von Forschern durchgeführt, um welche Art von Informationen zu erhalten? Und um Behandlungen oder Impfstoffe zu entwickeln?
Erklärungen von Dr. Chantal Snoeck, Virologin am Luxembourg Institute of Health (LIH).
Dr. Snoeck, Sie arbeiten am LIH unter anderem am H1N1-Schweinegrippevirus, das 2009 für eine weltweite Pandemie verantwortlich war. Wie identifizieren Wissenschaftler ein neues ansteckendes Virus wie das Coronavirus?
CS: Zur Identifizierung eines neuen Virus sind verschiedene Schritte möglich. Im Fall des neuen Coronavirus waren es vor allem epidemiologische Daten, die den Verdacht erweckten, dass etwas Abnormales vor sich ging: Mehrere in der Stadt Wuhan hospitalisierte Patienten litten an einer Lungenentzündung deren Ursache nicht bekannt war.
Die Unwirksamkeit von Antibiotika-Behandlungen und die bei Röntgenaufnahmen des Brustkorbs beobachteten Merkmale wiesen auf eine Lungenentzündung viralen und nicht bakteriellen Ursprungs hin. Basierend auf dieser Meldung hat eine epidemiologische Untersuchung betroffener Patienten ermöglicht, einen Fischmarkt als gemeinsamen Expositionsfaktor zwischen diesen verschiedenen Patienten zu identifizieren.
Und wie wird dann die genaue Identität des neuen Erregers ermittelt?
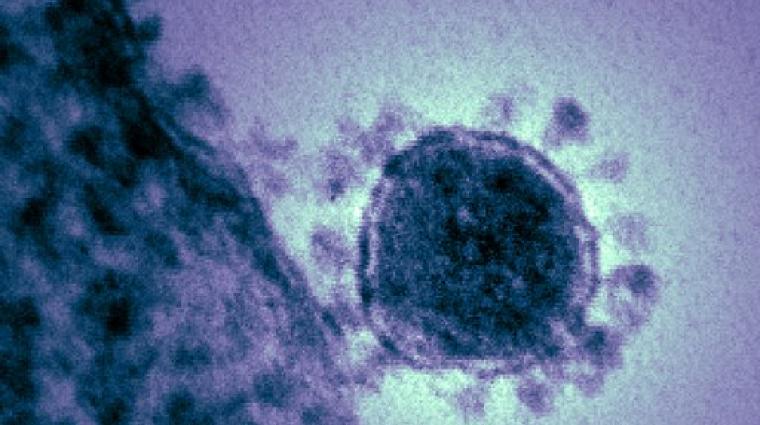
CS: Die Identität des Erregers kann zum einen durch Elektronenmikroskopie aufgeklärt werden: Die morphologischen Eigenschaften eines Virus geben Aufschluss über die Familie, zu der es gehört. Coronaviren haben ihren Namen von ihrer kugelförmigen Form, die von einer Krone umgeben ist - den Glykoproteinen, die in der Membran des Virus verankert sind und für das Andocken und den Eintritt des Virus in eine Zelle unerlässlich sind.
Bild: Eingefärbtes Transmissionselektronenmicrograph des Middle Eastern Respiratory Syndrom (MERS)-Coronavirus, der 2012 auftauchte.
In den letzten Jahren hat eine neue Technik die Vielzahl von Labortests zur Identifizierung und Charakterisierung eines Krankheitserregers ergänzt und die sogenannte "molekulare" Epidemiologie revolutioniert: Hochgeschwindigkeitssequenzierung - Next Generation Sequencing in englischer Sprache.
Aus einer Viruskultur oder sogar aus der Probe des Patienten ist es nun möglich, in wenigen Stunden die vollständige Sequenz des Virusgenoms zu erhalten. Für die Genomsequenzierung des SARS-Coronavirus in den Jahren 2002/2003 brauchte man noch einen Monat nach Identifizierung des Erregers. Das Genom des neuen Coronavirus war bereits am 10. Januar 2020 in der GISAID-Datenbank verfügbar, 10 Tage nach der Identifikation des neuen Virus.
Infobox
Die Sequenzierung von genetischem Material ist seit 1977 dank F. Sanger und Kollegen möglich, erfordert jedoch zunächst die Vermehrung eines kleinen Teils des Genoms unter Verwendung spezifischer Primer, dh kleiner DNA-Stränge, deren Sequenz bekannt ist und welche komplementär zum Ziel-DNA-Fragment sind. Diese Technik begrenzt daher sowohl die Größe der sequenzierten Fragmente als auch die Anzahl der erhaltenen Sequenzen und die Kenntnis des Genoms von vornherein. Und genau in diesen letzteren Aspekten hat die Hochgeschwindigkeitssequenzierung die Sequenzierung revolutioniert: Es ist keine A-priori-Information mehr erforderlich, und Forscher erhalten eine enorme Menge überlappender Sequenzen, um dann ein Genom durch Rekonstruktion mithilfe von Bioinformatik-Tools zu ermöglichen.
Das ist in der Tat beeindruckend. Wie wird dann der Verwandtschaftsgrad mit anderen Viren ermittelt?
CS: Die Genomsequenz des neuen Coronavirus wird mit dem Genom der anderen bekannten Coronaviren verglichen: Der Verwandtschaftsgrad, auch genetischer Abstand genannt, zwischen zwei Viren entspricht dem Prozentsatz identischer Basen zwischen diesen beiden Viren im gesamten Genom. Dieser wird mit statistischen Modellen berechnet, die die genetische Entwicklung von Organismen berücksichtigen.
Darüber hinaus ermöglichen phylogenetische Analysen die Verfolgung der Verwandtschafts- und Evolutionsbeziehungen zwischen genetischen Sequenzen, hier den Genomen verschiedener Coronaviren, dank der in den Sequenzen enthaltenen Signale. Diese Ergebnisse werden in Form eines Baumes dargestellt, der die wahrscheinlichen Zusammenhänge und die genetische Vorgeschichte zusammenfasst. Wir können das mit dem Aufbau eines Stammbaums vergleichen.
Welche Schlussfolgerungen zogen die Forscher in Bezug auf das neue Coronavirus?
Eine Woche nach der Veröffentlichung des Genoms des neuen Coronavirus war auch ein diagnostischer Test verfügbar. Wie entwickelt sich ein solcher diagnostischer Test?
CS: Heutzutage kann die Präsenz eines Virus in einer Probe durch die Polymerase Chain Reaction (PCR) schnell nachgewiesen werden. Diese Technik basiert auf der Amplifikation eines Teils des Genoms des Virus, wenn es in der Probe vorhanden ist, unter Verwendung von Primern, die spezifisch für das Genom des gesuchten Virus sind. Die rasche Veröffentlichung des 2019-nCoV-Genoms war für die rasche Entwicklung von PCRs zum Nachweis von Bedeutung.
Um einen solchen Test zu entwickeln, genügt es, eine Region im Genom des Virus zu finden, die bestimmte Kriterien erfüllt, beispielsweise, dass sie sich ausreichend von anderen Coronaviren unterscheidet, um sicherzustellen, dass der Test sie nicht ebenfalls erkennt. Die Identifizierung dieser Region erfolgt durch bioinformatische Tools, die die Sequenzen viraler Genome vergleichen. Anschließend wird der Test unter anderem validiert, indem Proben getestet werden, deren Infektionsstatus bekannt ist (positiv oder negativ für das Virus), und indem überprüft wird, ob andere verwandte Viren (andere Coronaviren) erkannt werden oder solche, die ähnliche Symptome verursachen (wie das Grippevirus).
Welche weiteren Analysen werden dann von den Forschern durchgeführt, um schnell weitere Informationen über das neue Virus zu erhalten?
CS: Anders als das Ebola-Virus, das weltweit in einem Labor der Sicherheitsstufe 4 in einer begrenzten Anzahl gehandhabt werden muss, kann das neue Coronavirus in einem Labor der Stufe 3 gehandhabt werden, was seine experimentelle Charakterisierung erleichtert. Nach seiner Entdeckung muss das Virus in vitro (im Labor, außerhalb eines lebenden Organismus) und in vivo (in lebenden Organismen) so gut wie möglich charakterisiert werden, um es zu verstehen.
Die Zellen, die das Virus befällt, können bestimmt werden indem versucht wird, eine Gruppe von Zelllinien tierischen und menschlichen Ursprungs zu infizieren. Es ermöglicht auch die Untersuchung des potentiellen Wirtsspektrums des Virus, um seinen potentiellen Ursprung sowie andere Tiere zu bestimmen, die den Übertragungszyklus des Virus wahrscheinlich aufrechterhalten.

Dieses Foto aus dem Jahr 2005 zeigt einen Mikrobiologen des Center for Disease Control and Prevention, der ein Influenzavirus untersucht, das in einer kalibrierten Flasche mit überstehendem Kulturmedium enthalten ist. Das Bild wurde in einer Laborumgebung der Biosicherheitsstufe 3 aufgenommen, in der Wissenschaftler unter einer Strömungshaube arbeiten, wobei die Luft außerhalb der Haube innerhalb der Grenzen der Haube angesaugt und dann Krankheitserreger herausgefiltert werden, bevor die Luft in die autonome Laboratmosphäre zurückgeführt wird. Die aktuellen Empfehlung für die Arbeit mit dem neuen Coronavirus ist die Biosicherheitsstufe 2, mit Ausnahme der IIsolierung des Virus in Zellkultur und der anfänglichen Charakterisierung der in den Kulturen von gewonnenen Viren aus '2019-nCoV-Proben, welche in der Biosicherheitsstufe 3 durchgeführt werden müssen. Foto: James Gathany Inhalteanbieter: CDC
Und auf molekularer Ebene?
CS: Der zelluläre Rezeptor, dh das Molekül, mit dem das Virus interagiert, um in die Zielzelle eindringen zu können, wurde ebenfalls bereits in vitro identifiziert. Dieses Wissen ermöglicht es dann der potentiellen Entwicklung von Molekülen, ihren Eintritt in die Zelle zu blockieren. In diesem Zusammenhang ist die Bioinformatik auch ein wesentliches Instrument zur Charakterisierung von Krankheitserregern geworden. Eine Modellanalyse der Glykoproteinstruktur des neuen Coronavirus basierend auf dem SARS-Virus legt beispielsweise nahe, dass der zelluläre Rezeptor für das Virus mit dem des SARS-Virus sowie anderen Coronaviren identisch sein könnte.
Das Verständnis, wie sich das Virus in der Zelle kopiert, ist auch wichtig für die Entwicklung eines Moleküls, das die Replikation (das Kopieren des Erbguts) verringern könnte oder dass das Virus wieder aus der infizierten Zelle austreten kann, wie dies bei Oseltamivir für das Influenzavirus der Fall ist. Ein weiterer möglicher Durchbruch ist die Entwicklung eines monoklonalen Antikörpers gegen das Virus, wie Palivizumab, der zur Vorbeugung schwerer Fälle von respiratorischem Syncytialvirus (RSV) bei sehr kleinen Kindern verabreicht werden kann.
Es scheint, dass vieles bereits im Labor oder mit dem Computer bestimmt werden kann. Sind Tierversuche dann noch nötig?
CS: Tierversuche bleiben wichtig, insbesondere um den genauen Übertragungsmodus eines Virus, seinen Übertragungsgrad zwischen einem infizierten Tier und einem gesunden Tier, die Faktoren, die die Schwere der Infektion beeinflussen können, zu verstehen oder um experimentelle Behandlungen vor klinischen Studien am Menschen zu testen.
Im Fall des Influenzavirus beispielsweise wird das Frettchen als experimentelles Modell angesehen, das die Übertragungsart des Virus reproduziert, und wird zur Beurteilung der Kapazität verwendet. Tiermodelle für Coronaviren wie SARS und MERS sind jedoch schwierig zu etablieren und haben Einschränkungen.
Parallel dazu müssen sero-epidemiologische Studien an Tieren (die bestimmen, welches Tier Antikörper gegen dieses bestimmte Virus aufweist) In-vitro- / In-vivo-Studien ergänzen, um das Ausmaß des Wirtsspektrums des Virus zu verstehen. Solche Studien am Menschen, bei Leuten, die häufig Kontakt mit ihren Tieren haben, werden auch feststellen, ob frühere und unentdeckte Infektionen aufgetreten sein können.
Werden Behandlungsoptionen oder Impfstoffe auch sofort getestet oder entwickelt?
CS: Angesichts der Ähnlichkeit der Symptome, die durch SARS und 2019-nCoV ausgelöst werden und basierend auf existierenden antiviralen Medikamenten haben Wissenschaftler dieses Wissen bereits zur experimentellen Behandlung bestimmter mit 2019-nCoV infizierter Patienten mit Lopinavir und Ritonavir verwendet. Lopinavir und Ritonavir sind Proteasehemmer, die insbesondere bei der Behandlung von HIV eingesetzt werden. Andere experimentelle Behandlungen, die gegen das SARS- oder MERS-Coronavirus entwickelt wurden, könnten möglicherweise zur Bekämpfung des neuen Coronavirus beitragen.
Schneller als je zuvor wurde ebenfalls bereits mit der Entwicklung von Impfstoffen begonnen. Drei Projekte, die auf drei verschiedenen Methoden basieren, werden bereits finanziell unterstützt. Die Verfügbarkeit der viralen Genomsequenz trug erneut zum raschen Start experimenteller Studien bei, die mit technologischen Fortschritten bei rekombinanten Impfstoffen einhergingen. Die Suche nach einem Impfstoff gegen MERS-CoV wird auch das Verfahren zur Erlangung eines Impfstoffs gegen 2019-nCoV beschleunigen.
Was sind die nächsten Schritte für Forscher, die an dem neuen Coronavirus arbeiten?
- Verständnis der genauen Übertragungsart: durch Tröpfchen (Ansteckung durch engen Kontakt) oder durch Aerosole (Ansteckung zwischen zwei weiter entfernten Personen)
- Wenn man das Spektrum der durch das Virus ausgelösten Symptome versteht, ist der tatsächliche Anteil milder, sogar asymptomatischer Fälle im Vergleich zu schwereren Fällen leichter zu identifizieren
- Die Suche nach dem Tierreservoir des Virus
- Bestimmung des Wirtsspektrums
- Grundlegendes zum genauen Replikationsmodus des Virus
- Die Suche nach einer wirksamen Behandlung
- Entwicklung eines Impfstoffs
Dr. Snoeck, vielen Dank für diese sehr detaillierten Informationen!
Chantal Snoeck ist Virologin im Team für klinische und angewandte Virologie in der Abteilung für Infektion und Immunität am LIH. Die Aktivitäten der Gruppe umfassen Überwachung, Diagnose, epidemiologische Untersuchung, molekulare Epidemiologie und Immunität gegen verschiedene Viruserkrankungen wie Masern. Informationen über die Prävalenz, Variabilität, Pathogenität der Viren und den Grad der Immunität der Bevölkerung sind für Entscheidungsträger im Bereich der öffentlichen Gesundheit wichtig, um Infektionskrankheiten zu kontrollieren und zu behandeln. Chantal Snoeck arbeitet insbesondere an zoonotischen Viruserkrankungen wie der Vogel- und Schweinegrippe, dem Hepatitis-E-Virus sowie viralen Tierkrankheiten mit wichtigen wirtschaftlichen Folgen.
Text: Chantal Snoeck (LIH)
Interviewfragen und Redaktion: Michèle Weber (FNR)
Foto: National Institute of Allergy and Infectious Diseases(NIAID) und Public Health Image Library des CDC via Wikimedia Commons.
Infobox
Gralinski and Menachery. Return of the coronaviruses: 2019-nCoV. Viruses 2020.